近日,伦敦大学学院Helen C. Hailes教授团队在《Green Chemistry》上发表了题为“The discovery of new metagenomic urethanases utilising a novel colorimetric assay for applications in the biodegradation of polyurethanes”的研究论文。


该研究基于家庭排水系统宏基因组资源,成功挖掘出三种此前未报道的酰胺酶,并开发了一种基于酪氨酸酶(RsTYR)氧化反应的高通量颜色测定法,可快速筛选具聚氨酯降解能力的酶类。研究结果表明,这些新型酶能有效水解以亚甲基二苯胺(MDA)为骨架的聚氨酯模型底物,并在经过球磨与纤维素酶预处理的商业织物体系中,实现了弹性纤维组分的显著降解。该研究为聚氨酯及其衍生纤维材料的生物降解与回收提供了新的酶学工具,并提出了可扩展至其他难降解高分子的高效筛选与验证策略。
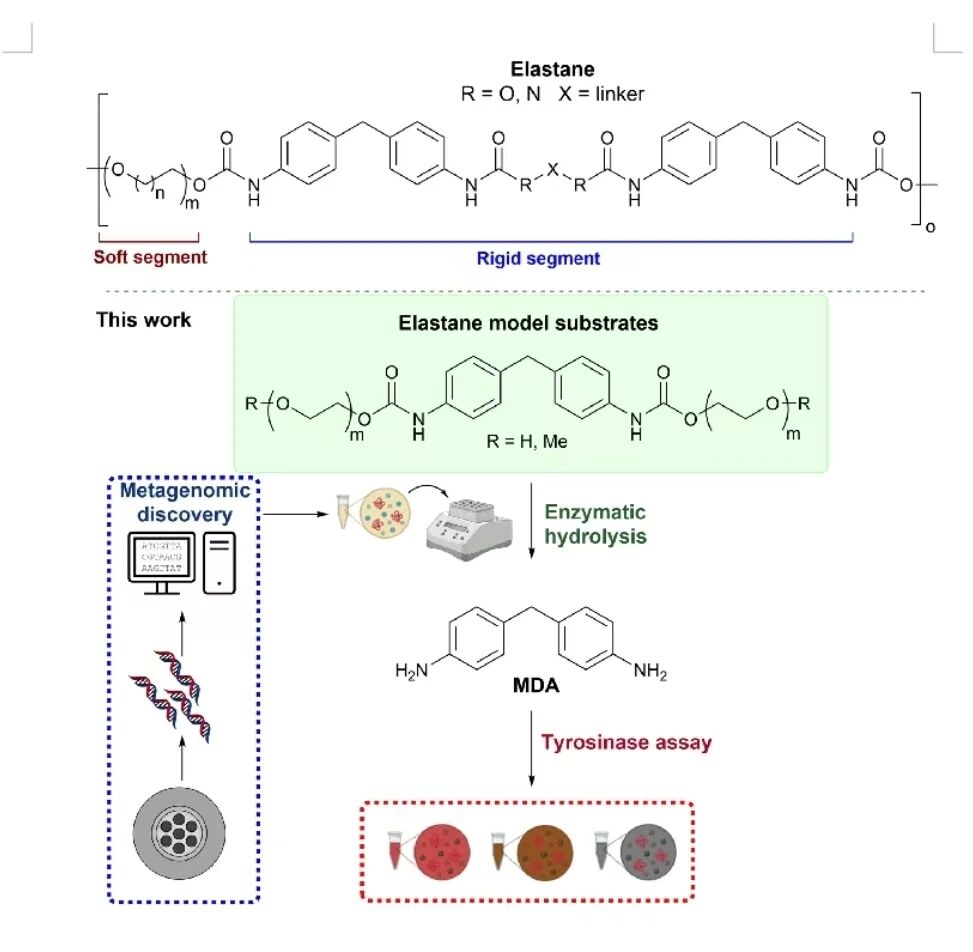
研究背景
全球塑料污染问题日益严峻,其中纺织品废弃物年产生量高达9200万吨,大部分最终被填埋或进入环境。弹性纤维(Spandex)是聚氨酯(PU)类材料的典型代表,其分子结构由芳香族“硬段”(多以4,4'-亚甲基二苯胺MDA为核心)和柔性的聚醚或聚酯“软段”,这种“软硬段”嵌段结构赋予材料优异的弹性与韧性,导致其在自然条件下极难降解。与PET等聚酯类塑料相比,聚氨酯的生物降解研究仍处于起步阶段,尤其是能断裂氨基甲酸酯键(–NH–COO–)的酰胺酶(urethanase)极为稀少。为此,研究团队以家庭排水系统这一富含蛋白质残渣和多样微生物群的环境为样本来源,构建宏基因组数据库,并设计一种酪氨酸酶辅助的显色测定体系,以实现对潜在聚氨酯水解酶的快速、高通量筛选与验证。
图文导读
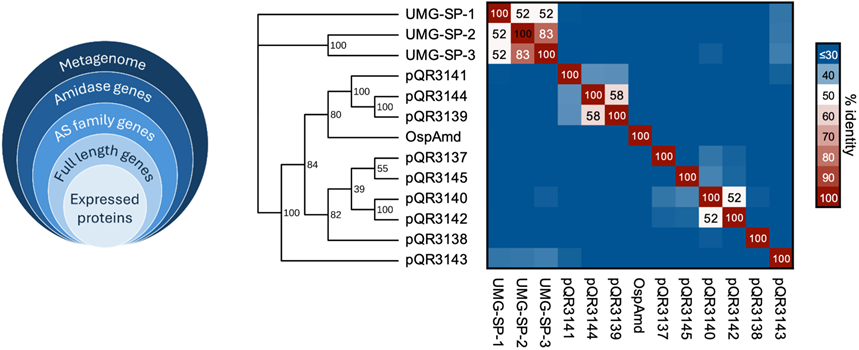
图1 从属于AS 家族的排水宏基因组中新鉴定的酰胺酶(左)。检索到的酰胺酶(包括文献中报道的氨基甲酸酯酶)的单位矩阵百分比和相应的分支图。
研究团队利用自建的排水系统宏基因组数据库(富含蛋白质残渣与微生物群),通过Pfam数据库搜索含amidase signature(AS)结构域(PF01425)的开放阅读框(ORF)。共识别出46条潜在序列,筛选得到9个完整序列并验证其具备典型的Ser–Ser–Lys催化三联体(pQR3137–pQR3145)。这些基因被克隆至pET-29a(+)载体中表达,随后进行系统发育分析。结果显示,这些新型酶与已报道的UMG-SP系列氨基甲酸酯酶在系统发育树上形成独立分支,提示其可能具备新颖的底物识别位点与催化特征。
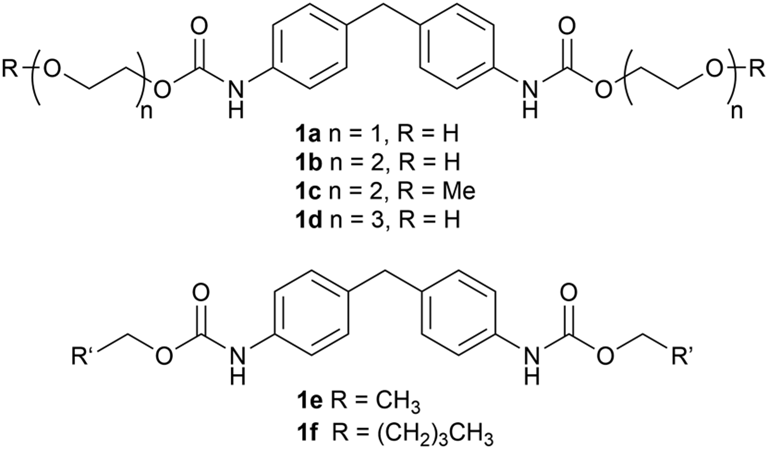
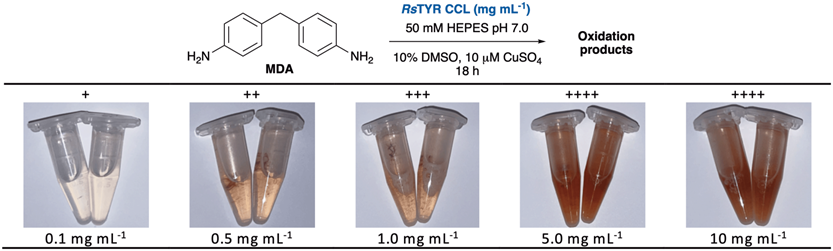
图2 (A)弹性纤维模型基质1a-1f (B)开发 MDA 氧化的 RsTYR 测定法。酶的浓度表示为总重悬浮冻干 CCL 粉末。
研究团队建立了一种基于MDA释放与显色反应的快速检测体系:酰胺酶水解MDA基模型底物(1a–1f)后释放出MDA,随后由RsTYR氧化生成红色醌类化合物,从而实现酶活的直接显色检测。实验优化后发现,在含 5 mg/mL 细胞裂解液的体系中,RsTYR 能在 18 小时内实现明显显色,形成高灵敏度的检测反应。相比传统HPLC定量,该方法简便、重复性高,且可在96孔板中并行操作,适合高通量筛选新型聚氨酯水解酶。
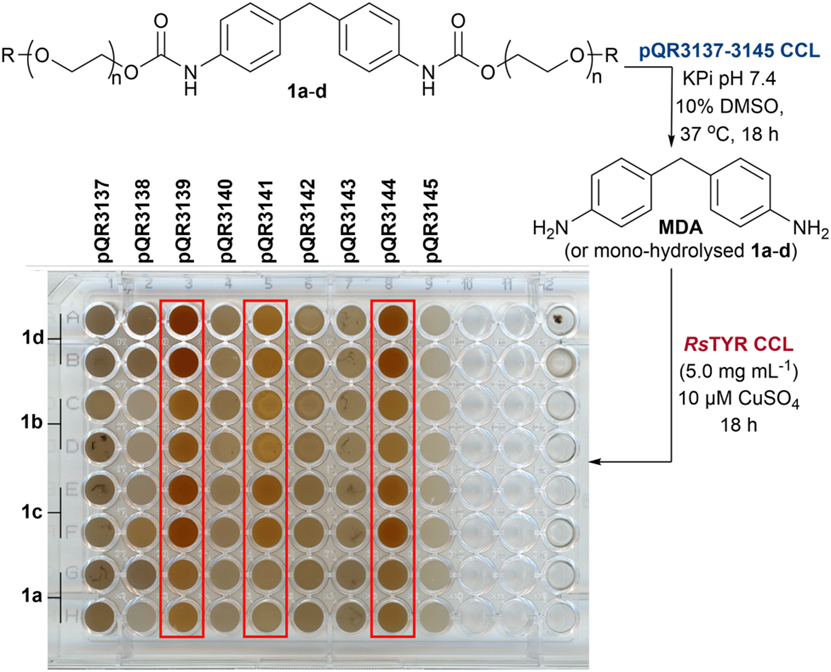
图 3 使用颜色测定法初筛宏基因组酰胺酶对模型底物(1a-1d)的水解活性,pQR3139, pQR3141, pQR3144显示阳性(红色)。
9种候选酶均在E. coli BL21中表达为粗酶液,利用显色筛选其对模型底物1a–1d的水解活性。结果显示,pQR3139、pQR3141和pQR3144 三种样品在反应后均出现显著红色变化,表明其具备酰胺酶活性。进一步HPLC定量分析表明,这三种酶可有效水解PEG链长度递增的模型底物,且活性随PEG链长增加而增强, 说明其在疏水性底物识别上具显著优势。
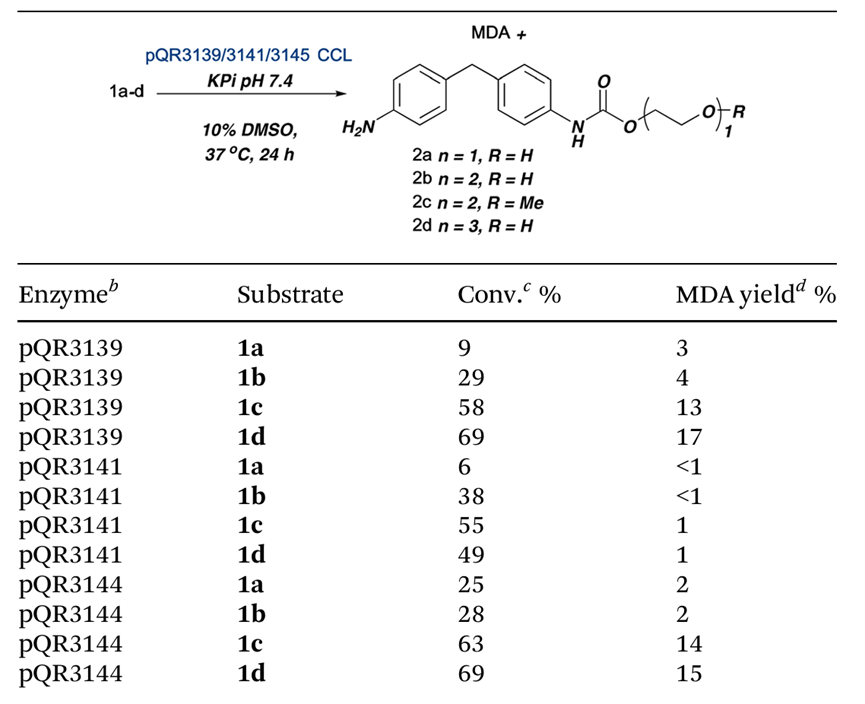
表 1 通过HPLC定量三种候选酶对模型底物(1a-1d)的转化情况及MDA产率
底物1c和1d的水解转化率为55–69%,生成MDA;1a和1b的转化率较低(30–38%),产物以单水解中间体为主。结果与比色检测一致,验证了显色体系的准确性与可靠性。研究指出,底物链长与柔性程度显著影响酶的底物适配性。
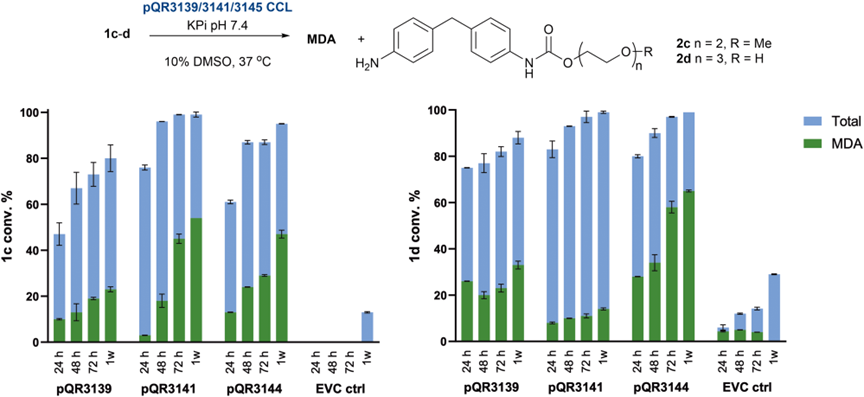
图4 与pQR3139、3141和3144进行1cd的时间点反应。使用酶作为CCL,总蛋白浓度为20 mg mL−1,酶含量为10%,反应进行24、48和72小时和1周,重复进行。
以1c和1d为底物,延长反应时间至7天,三种酶均表现出持续的水解能力,生成MDA及中间体(2a–2d)。其中,pQR3144在反应一周后MDA产率最高(65%),表明其稳定性和催化效率优于其他两种酶。
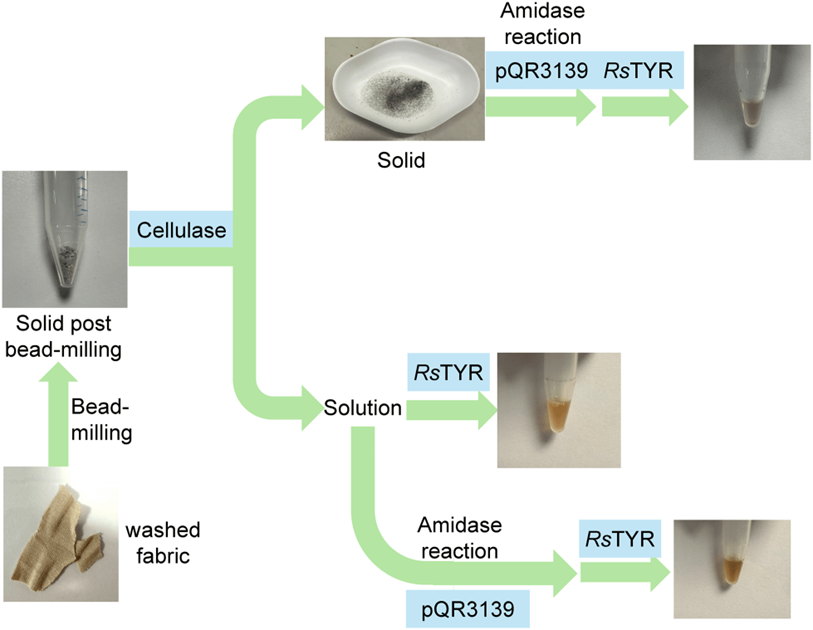
图 5 商业织物(粘胶/弹性纤维、棉/弹性纤维)酶法降解工作流程,包括洗涤、球磨、纤维素酶预处理、酰胺酶处理及颜色测定。
为验证其应用潜力,研究选择含弹性纤维的商业织物。通过球磨(添加甲醇以增强纤维破碎)及纤维素酶预处理,破坏织物结构、去除纤维素基质,为酰胺酶降解聚氨酯段创造条件。
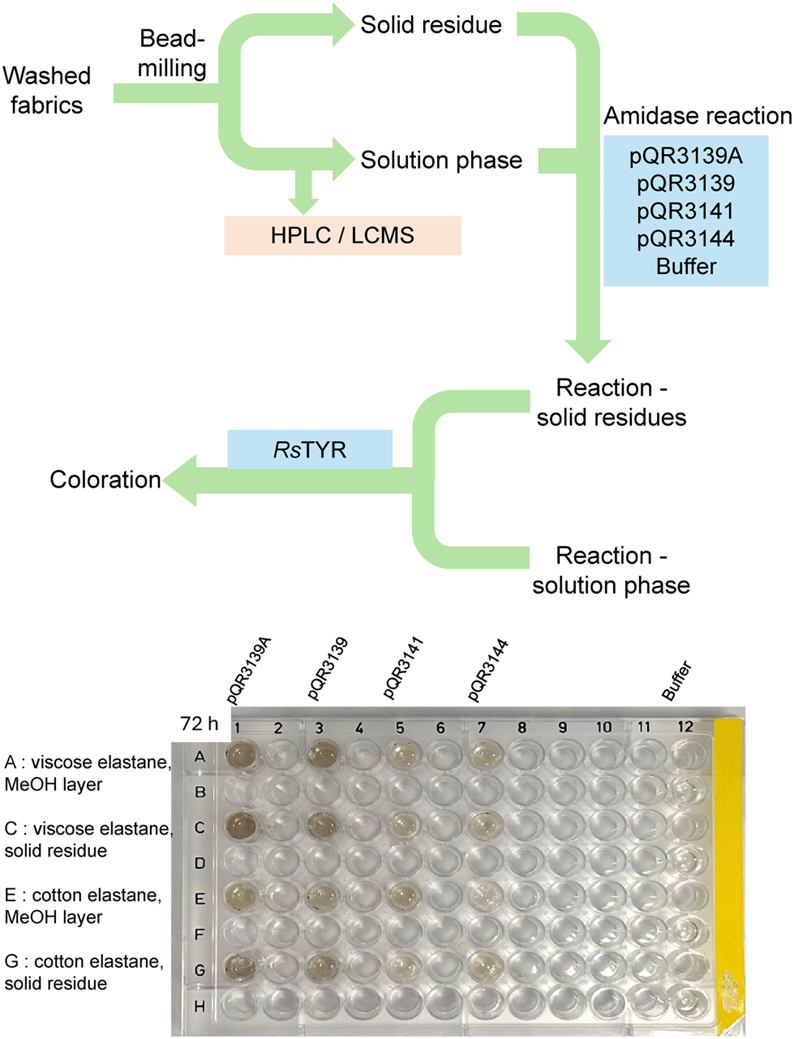
图 6 经pQR3139处理后的织物提取液在TYR测定中呈现阳性显色反应(红色),表明弹性纤维发生降解。
使用活性最高的pQR3139处理后,织物提取液经RsTYR显色检测呈明显红橙色,而阴性对照无变化,表明聚氨酯组分被有效降解并释放出MDA。¹H NMR分析进一步检测到芳香环与甲撑桥信号峰,为降解反应提供直接证据。
主要结论
本研究通过整合宏基因组挖掘、酶学筛选与应用验证,系统地实现了聚氨酯降解酰胺酶的发现与功能鉴定。研究团队从家庭排水系统宏基因组中成功挖掘出三种新型酰胺酶(pQR3139、pQR3141、pQR3144),并证实其对MDA型聚氨酯模型底物具有显著水解活性;同时,开发出基于酪氨酸酶氧化显色反应的高通量颜色测定法,实现了酰胺酶活性的快速筛选与定性检测。进一步地,研究在经球磨与纤维素酶预处理的商业织物体系中验证了这些酶的降解能力,成功实现了聚氨酯弹性纤维的生物降解。该成果不仅拓展了聚氨酯及其他难降解高分子材料的生物降解酶资源库,也建立了“宏基因组挖掘—酶显色筛选—实体降解验证”的一体化研究策略,为未来通过酶工程与代谢途径优化提升聚氨酯降解效率提供了新方向,并为混合纺织品的分子级循环利用奠定了重要基础。
更多最新塑料行情、最全塑料网资讯尽在德富塑料网! (原乐从塑料网,始于2002年,是基于广东乐从德富塑料城搭建的, 华南塑化行业首选塑料原料、塑料制品、 塑料机械一站式采购平台。更新、更全的塑料行情、塑料资讯尽在德富塑料网。

(德富塑料网公众号)
免责声明:本文来自于网络,资源仅供参考,如有侵犯了您的权益请立即与我们联系!我们将及时撤除。